نقاط بيانات اللون المشروط خارج نطاقات الثقة في R
-
30-09-2019 - |
سؤال
أحتاج إلى تلوين نقاط البيانات الموجودة خارج نطاقات الثقة على المؤامرة أدناه بشكل مختلف عن تلك الموجودة في النطاقات. هل يجب علي إضافة عمود منفصل إلى مجموعة البيانات الخاصة بي لتسجيل ما إذا كانت نقاط البيانات ضمن نطاقات الثقة؟ هل يمكنك تقديم مثال من فضلك؟
مثال مجموعة البيانات:
## Dataset from http://www.apsnet.org/education/advancedplantpath/topics/RModules/doc1/04_Linear_regression.html
## Disease severity as a function of temperature
# Response variable, disease severity
diseasesev<-c(1.9,3.1,3.3,4.8,5.3,6.1,6.4,7.6,9.8,12.4)
# Predictor variable, (Centigrade)
temperature<-c(2,1,5,5,20,20,23,10,30,25)
## For convenience, the data may be formatted into a dataframe
severity <- as.data.frame(cbind(diseasesev,temperature))
## Fit a linear model for the data and summarize the output from function lm()
severity.lm <- lm(diseasesev~temperature,data=severity)
# Take a look at the data
plot(
diseasesev~temperature,
data=severity,
xlab="Temperature",
ylab="% Disease Severity",
pch=16,
pty="s",
xlim=c(0,30),
ylim=c(0,30)
)
title(main="Graph of % Disease Severity vs Temperature")
par(new=TRUE) # don't start a new plot
## Get datapoints predicted by best fit line and confidence bands
## at every 0.01 interval
xRange=data.frame(temperature=seq(min(temperature),max(temperature),0.01))
pred4plot <- predict(
lm(diseasesev~temperature),
xRange,
level=0.95,
interval="confidence"
)
## Plot lines derrived from best fit line and confidence band datapoints
matplot(
xRange,
pred4plot,
lty=c(1,2,2), #vector of line types and widths
type="l", #type of plot for each column of y
xlim=c(0,30),
ylim=c(0,30),
xlab="",
ylab=""
)
المحلول
أسهل طريقة ربما لحساب متجه TRUE/FALSE القيم التي تشير إذا كانت نقطة البيانات داخل فاصل الثقة أم لا. سأقوم بإعادة تحديد مثالك قليلاً حتى يتم إكمال جميع الحسابات قبل تنفيذ أوامر التخطيط- وهذا يوفر فصلًا نظيفًا في منطق البرنامج الذي يمكن استغلاله إذا كنت تريد حزم بعض هذا في وظيفة .
الجزء الأول هو نفسه إلى حد كبير ، إلا أنني استبدلت المكالمة الإضافية إلى lm() داخل predict() مع ال severity.lm المتغير- ليست هناك حاجة لاستخدام موارد حوسبة إضافية لإعادة حساب النموذج الخطي عندما يكون لدينا بالفعل تخزينه:
## Dataset from
# apsnet.org/education/advancedplantpath/topics/
# RModules/doc1/04_Linear_regression.html
## Disease severity as a function of temperature
# Response variable, disease severity
diseasesev<-c(1.9,3.1,3.3,4.8,5.3,6.1,6.4,7.6,9.8,12.4)
# Predictor variable, (Centigrade)
temperature<-c(2,1,5,5,20,20,23,10,30,25)
## For convenience, the data may be formatted into a dataframe
severity <- as.data.frame(cbind(diseasesev,temperature))
## Fit a linear model for the data and summarize the output from function lm()
severity.lm <- lm(diseasesev~temperature,data=severity)
## Get datapoints predicted by best fit line and confidence bands
## at every 0.01 interval
xRange=data.frame(temperature=seq(min(temperature),max(temperature),0.01))
pred4plot <- predict(
severity.lm,
xRange,
level=0.95,
interval="confidence"
)
الآن ، سنحسب فترات الثقة لنقاط البيانات الأصلية ونقوم بإجراء اختبار لمعرفة ما إذا كانت النقاط داخل الفاصل الزمني:
modelConfInt <- predict(
severity.lm,
level = 0.95,
interval = "confidence"
)
insideInterval <- modelConfInt[,'lwr'] < severity[['diseasesev']] &
severity[['diseasesev']] < modelConfInt[,'upr']
ثم سنقوم بالمؤامرة- أولاً وظيفة التخطيط عالية المستوى plot(), ، كما استخدمته في مثالك ، لكننا سنرسم النقاط داخل الفاصل الزمني فقط. سنتابع بعد ذلك وظيفة المستوى المنخفض points() والتي سوف ترسم جميع النقاط خارج الفاصل الزمني بلون مختلف. أخيراً، matplot() سيتم استخدامها لملء فترات الثقة كما استخدمتها. ولكن بدلا من الاتصال par(new=TRUE) أفضل تمرير الحجة add=TRUE إلى وظائف عالية المستوى لجعلها تتصرف مثل وظائف المستوى المنخفض.
استخدام par(new=TRUE) يشبه لعب خدعة قذرة وظيفة التخطيط- والتي يمكن أن يكون لها عواقب غير متوقعة. ال add يتم توفير الوسيطة من قبل العديد من الوظائف التي تجعلها تضيف معلومات إلى مؤامرة بدلاً من إعادة رسمها- أوصي باستغلال هذه الوسيطة كلما أمكن ذلك وأتراجع par() التلاعب كملاذ أخير.
# Take a look at the data- those points inside the interval
plot(
diseasesev~temperature,
data=severity[ insideInterval,],
xlab="Temperature",
ylab="% Disease Severity",
pch=16,
pty="s",
xlim=c(0,30),
ylim=c(0,30)
)
title(main="Graph of % Disease Severity vs Temperature")
# Add points outside the interval, color differently
points(
diseasesev~temperature,
pch = 16,
col = 'red',
data = severity[ !insideInterval,]
)
# Add regression line and confidence intervals
matplot(
xRange,
pred4plot,
lty=c(1,2,2), #vector of line types and widths
type="l", #type of plot for each column of y
add = TRUE
)
نصائح أخرى
حسنًا ، اعتقدت أن هذا سيكون سهلاً للغاية مع GGPLOT2 ، لكنني أدرك الآن أنه ليس لدي أي فكرة عن كيفية حساب حدود الثقة لـ STAT_SMOTH/GEOM_SMOOTH.
النظر في ما يلي:
library(ggplot2)
pred <- as.data.frame(predict(severity.lm,level=0.95,interval="confidence"))
dat <- data.frame(diseasesev,temperature,
in_interval = diseasesev <=pred$upr & diseasesev >=pred$lwr ,pred)
ggplot(dat,aes(y=diseasesev,x=temperature)) +
stat_smooth(method='lm') + geom_point(aes(colour=in_interval)) +
geom_line(aes(y=lwr),colour=I('red')) + geom_line(aes(y=upr),colour=I('red'))
هذا ينتج:alt text http://ifellows.ucsd.edu/pmwiki/uploads/main/strangeplot.jpg
لا أفهم لماذا لا يتماشى نطاق الثقة المحسوب بواسطة STAT_Smooth مع النطاق المحسوب مباشرة من التنبؤ (أي الخطوط الحمراء). هل يستطيع اي شخص ان يسلط الضوء على هذا؟
يحرر:
اكتشفه. يستخدم GGPLOT2 خطأ قياسي 1.96 * لرسم الفواصل الزمنية لجميع طرق التجانس.
pred <- as.data.frame(predict(severity.lm,se.fit=TRUE,
level=0.95,interval="confidence"))
dat <- data.frame(diseasesev,temperature,
in_interval = diseasesev <=pred$fit.upr & diseasesev >=pred$fit.lwr ,pred)
ggplot(dat,aes(y=diseasesev,x=temperature)) +
stat_smooth(method='lm') +
geom_point(aes(colour=in_interval)) +
geom_line(aes(y=fit.lwr),colour=I('red')) +
geom_line(aes(y=fit.upr),colour=I('red')) +
geom_line(aes(y=fit.fit-1.96*se.fit),colour=I('green')) +
geom_line(aes(y=fit.fit+1.96*se.fit),colour=I('green'))
أحببت الفكرة وحاولت جعل وظيفة لذلك. بالطبع إنه بعيد عن كونه مثاليًا. تعليقاتك مرحب بها
diseasesev<-c(1.9,3.1,3.3,4.8,5.3,6.1,6.4,7.6,9.8,12.4)
# Predictor variable, (Centigrade)
temperature<-c(2,1,5,5,20,20,23,10,30,25)
## For convenience, the data may be formatted into a dataframe
severity <- as.data.frame(cbind(diseasesev,temperature))
## Fit a linear model for the data and summarize the output from function lm()
severity.lm <- lm(diseasesev~temperature,data=severity)
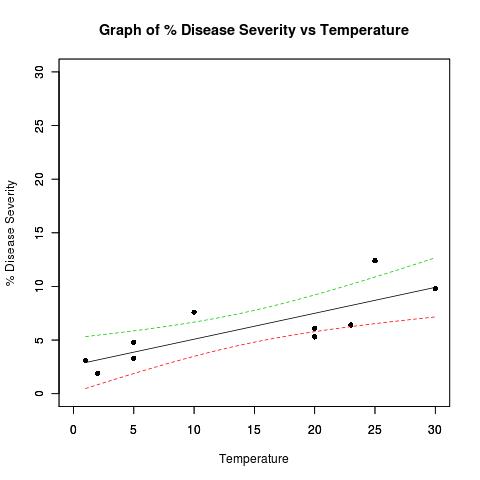
# Function to plot the linear regression and overlay the confidence intervals
ci.lines<-function(model,conf= .95 ,interval = "confidence"){
x <- model[[12]][[2]]
y <- model[[12]][[1]]
xm<-mean(x)
n<-length(x)
ssx<- sum((x - mean(x))^2)
s.t<- qt(1-(1-conf)/2,(n-2))
xv<-seq(min(x),max(x),(max(x) - min(x))/100)
yv<- coef(model)[1]+coef(model)[2]*xv
se <- switch(interval,
confidence = summary(model)[[6]] * sqrt(1/n+(xv-xm)^2/ssx),
prediction = summary(model)[[6]] * sqrt(1+1/n+(xv-xm)^2/ssx)
)
# summary(model)[[6]] = 'sigma'
ci<-s.t*se
uyv<-yv+ci
lyv<-yv-ci
limits1 <- min(c(x,y))
limits2 <- max(c(x,y))
predictions <- predict(model, level = conf, interval = interval)
insideCI <- predictions[,'lwr'] < y & y < predictions[,'upr']
x_name <- rownames(attr(model[[11]],"factors"))[2]
y_name <- rownames(attr(model[[11]],"factors"))[1]
plot(x[insideCI],y[insideCI],
pch=16,pty="s",xlim=c(limits1,limits2),ylim=c(limits1,limits2),
xlab=x_name,
ylab=y_name,
main=paste("Graph of ", y_name, " vs ", x_name,sep=""))
abline(model)
points(x[!insideCI],y[!insideCI], pch = 16, col = 'red')
lines(xv,uyv,lty=2,col=3)
lines(xv,lyv,lty=2,col=3)
}
استخدمه مثل هذا:
ci.lines(severity.lm, conf= .95 , interval = "confidence")
ci.lines(severity.lm, conf= .85 , interval = "prediction")