باستخدام R.Zoo لرسم سلسلة متعددة مع أشرطة الخطأ
-
26-09-2019 - |
سؤال
لدي بيانات تشبه هذا:
> head(data)
groupname ob_time dist.mean dist.sd dur.mean dur.sd ct.mean ct.sd
1 rowA 0.3 61.67500 39.76515 43.67500 26.35027 8.666667 11.29226
2 rowA 60.0 45.49167 38.30301 37.58333 27.98207 8.750000 12.46176
3 rowA 120.0 50.22500 35.89708 40.40000 24.93399 8.000000 10.23363
4 rowA 180.0 54.05000 41.43919 37.98333 28.03562 8.750000 11.97061
5 rowA 240.0 51.97500 41.75498 35.60000 25.68243 28.583333 46.14692
6 rowA 300.0 45.50833 43.10160 32.20833 27.37990 12.833333 14.21800
كل مجموعة GroupName هي سلسلة بيانات. بما أنني أرغب في رسم كل سلسلة بشكل منفصل ، فقد قمت بفصلها على هذا النحو:
> A <- zoo(data[which(groupname=='rowA'),3:8],data[which(groupname=='rowA'),2])
> B <- zoo(data[which(groupname=='rowB'),3:8],data[which(groupname=='rowB'),2])
> C <- zoo(data[which(groupname=='rowC'),3:8],data[which(groupname=='rowC'),2])
إيتا:
Thanks to gd047: Now I'm using this:
z <- dlply(data,.(groupname),function(x) zoo(x[,3:8],x[,2]))
تبدو كائنات حديقة الحيوان الناتجة هكذا:
> head(z$rowA)
dist.mean dist.sd dur.mean dur.sd ct.mean ct.sd
0.3 61.67500 39.76515 43.67500 26.35027 8.666667 11.29226
60 45.49167 38.30301 37.58333 27.98207 8.750000 12.46176
120 50.22500 35.89708 40.40000 24.93399 8.000000 10.23363
180 54.05000 41.43919 37.98333 28.03562 8.750000 11.97061
240 51.97500 41.75498 35.60000 25.68243 28.583333 46.14692
300 45.50833 43.10160 32.20833 27.37990 12.833333 14.21800
لذلك إذا كنت أرغب في رسم dist.mean مع الزمن وتشمل أشرطة خطأ تساوي +/- dist.sd لكل سلسلة:
- كيف يمكنني الجمع بين A و B و C Dist.mean و Dist.sd؟
- كيف أقوم بعمل مؤامرة بار ، أو ربما أفضل, ، رسم بياني خط للكائن الناتج؟
المحلول
لا أرى الهدف من تقسيم البيانات إلى ثلاث قطع فقط لأضطر إلى الجمع بينها للمخطط. هنا مؤامرة باستخدام ggplot2 مكتبة:
library(ggplot2)
qplot(ob_time, dist.mean, data=data, colour=groupname, geom=c("line","point")) +
geom_errorbar(aes(ymin=dist.mean-dist.sd, ymax=dist.mean+dist.sd))
هذا يمسح قيم الوقت على طول المقياس الطبيعي ، يمكنك استخدامه scale_x_continuous لتحديد العلامات الدعائية في قيم الوقت الفعلية. إن وجودها متباعدة بالتساوي أمر أصعب: يمكنك التحويل ob_time إلى عامل ، ولكن بعد ذلك qplot يرفض توصيل النقاط بخط.
الحل 1 - رسم بياني بار:
qplot(factor(ob_time), dist.mean, data=data, geom=c("bar"), fill=groupname,
colour=groupname, position="dodge") +
geom_errorbar(aes(ymin=dist.mean-dist.sd, ymax=dist.mean+dist.sd), position="dodge")
الحل 2 - أضف خطوطًا يدويًا باستخدام 1،2 ، ... إعادة ترميز العامل:
qplot(factor(ob_time), dist.mean, data=data, geom=c("line","point"), colour=groupname) +
geom_errorbar(aes(ymin=dist.mean-dist.sd, ymax=dist.mean+dist.sd)) +
geom_line(aes(x=as.numeric(factor(ob_time))))
نصائح أخرى
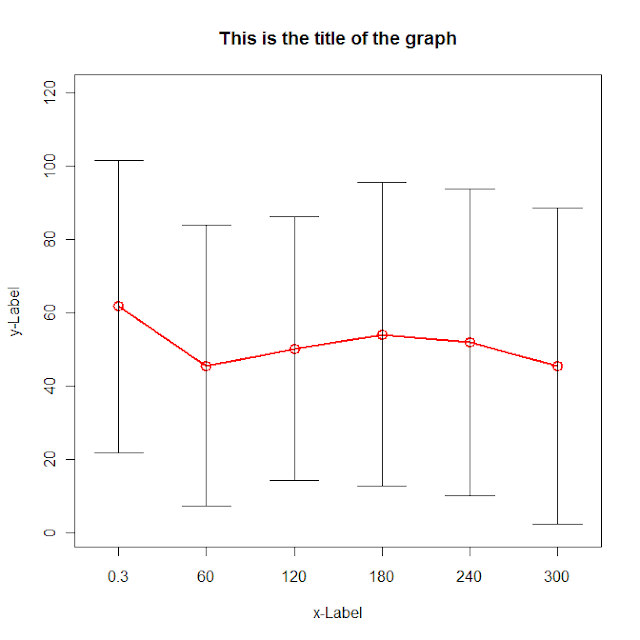
هذا تلميح للطريقة التي سأحاول القيام بها. لقد تجاهلت التجميع ، لذلك عليك تعديلها لتضمين أكثر من سلسلة واحدة. كما أنني لم أستخدم حديقة الحيوان لأنني لا أعرف الكثير.
g <- (nrow(data)-1)/(3*nrow(data))
plot(data[,"dist.mean"],col=2, type='o',lwd=2,cex=1.5, main="This is the title of the graph",
xlab="x-Label", ylab="y-Label", xaxt="n",
ylim=c(0,max(data[,"dist.mean"])+max(data[,"dist.sd"])),
xlim=c(1-g,nrow(data)+g))
axis(side=1,at=c(1:nrow(data)),labels=data[,"ob_time"])
for (i in 1:nrow(data)) {
lines(c(i,i),c(data[i,"dist.mean"]+data[i,"dist.sd"],data[i,"dist.mean"]-data[i,"dist.sd"]))
lines(c(i-g,i+g),c(data[i,"dist.mean"]+data[i,"dist.sd"], data[i,"dist.mean"]+data[i,"dist.sd"]))
lines(c(i-g,i+g),c(data[i,"dist.mean"]-data[i,"dist.sd"], data[i,"dist.mean"]-data[i,"dist.sd"]))
}
اقرأ البيانات في استخدام read.zoo مع الانقسام = وسيطة لتقسيمها بواسطة GroupName. ثم اربط معًا الخطوط السفلية والثانية. أخيرا ارسمهم.
Lines <- "groupname ob_time dist.mean dist.sd dur.mean dur.sd ct.mean ct.sd
rowA 0.3 61.67500 39.76515 43.67500 26.35027 8.666667 11.29226
rowA 60.0 45.49167 38.30301 37.58333 27.98207 8.750000 12.46176
rowA 120.0 50.22500 35.89708 40.40000 24.93399 8.000000 10.23363
rowA 180.0 54.05000 41.43919 37.98333 28.03562 8.750000 11.97061
rowB 240.0 51.97500 41.75498 35.60000 25.68243 28.583333 46.14692
rowB 300.0 45.50833 43.10160 32.20833 27.37990 12.833333 14.21800"
library(zoo)
# next line is only needed until next version of zoo is released
source("http://r-forge.r-project.org/scm/viewvc.php/*checkout*/pkg/zoo/R/read.zoo.R?revision=719&root=zoo")
z <- read.zoo(textConnection(Lines), header = TRUE, split = 1, index = 2)
# pick out the dist and sd columns binding dist with lower & upper
z.dist <- z[, grep("dist.mean", colnames(z))]
z.sd <- z[, grep("dist.sd", colnames(z))]
zz <- cbind(z = z.dist, lower = z.dist - z.sd, upper = z.dist + z.sd)
# plot using N panels
N <- ncol(z.dist)
ylab <- sub("dist.mean.", "", colnames(z.dist))
plot(zz, screen = 1:N, type = "l", lty = rep(1:2, N*1:2), ylab = ylab)
لا أعتقد أنك بحاجة إلى إنشاء كائنات حديقة حيوان لهذا النوع من المؤامرة ، سأفعل ذلك مباشرة من إطار البيانات. بالطبع ، قد تكون هناك أسباب أخرى لاستخدام كائنات حديقة الحيوان ، مثل الدمج الذكي ، التجميع ، إلخ.
خيار واحد هو segplot وظيفة من LatticeExtra
library(latticeExtra)
segplot(ob_time ~ (dist.mean + dist.sd) + (dist.mean - dist.sd) | groupname,
data = data, centers = dist.mean, horizontal = FALSE)
## and with the latest version of latticeExtra (from R-forge):
trellis.last.object(segments.fun = panel.arrows, ends = "both", angle = 90, length = .1) +
xyplot(dist.mean ~ ob_time | groupname, data, col = "black", type = "l")
باستخدام مجموعة بيانات Gabor التي تنتجها Gabor هذا:
