Usando R.Zoo para plotar várias séries com barras de erro
-
26-09-2019 - |
Pergunta
Eu tenho dados que se parecem com o seguinte:
> head(data)
groupname ob_time dist.mean dist.sd dur.mean dur.sd ct.mean ct.sd
1 rowA 0.3 61.67500 39.76515 43.67500 26.35027 8.666667 11.29226
2 rowA 60.0 45.49167 38.30301 37.58333 27.98207 8.750000 12.46176
3 rowA 120.0 50.22500 35.89708 40.40000 24.93399 8.000000 10.23363
4 rowA 180.0 54.05000 41.43919 37.98333 28.03562 8.750000 11.97061
5 rowA 240.0 51.97500 41.75498 35.60000 25.68243 28.583333 46.14692
6 rowA 300.0 45.50833 43.10160 32.20833 27.37990 12.833333 14.21800
Cada nome do grupo é uma série de dados. Como eu quero plotar cada série separadamente, eu as separei assim:
> A <- zoo(data[which(groupname=='rowA'),3:8],data[which(groupname=='rowA'),2])
> B <- zoo(data[which(groupname=='rowB'),3:8],data[which(groupname=='rowB'),2])
> C <- zoo(data[which(groupname=='rowC'),3:8],data[which(groupname=='rowC'),2])
ETA:
Thanks to gd047: Now I'm using this:
z <- dlply(data,.(groupname),function(x) zoo(x[,3:8],x[,2]))
Os objetos do zoológico resultantes são assim:
> head(z$rowA)
dist.mean dist.sd dur.mean dur.sd ct.mean ct.sd
0.3 61.67500 39.76515 43.67500 26.35027 8.666667 11.29226
60 45.49167 38.30301 37.58333 27.98207 8.750000 12.46176
120 50.22500 35.89708 40.40000 24.93399 8.000000 10.23363
180 54.05000 41.43919 37.98333 28.03562 8.750000 11.97061
240 51.97500 41.75498 35.60000 25.68243 28.583333 46.14692
300 45.50833 43.10160 32.20833 27.37990 12.833333 14.21800
Portanto, se eu quiser plotar dist. mean contra o tempo e incluir barras de erro iguais a +/- dist.sd para cada série:
- Como combino A, B, C Dist.Mean e Dist.SD?
- Como faço para fazer uma trama de bar, ou talvez melhor, um gráfico de linha do objeto resultante?
Solução
Não vejo o ponto de dividir os dados em três peças, apenas para combiná -los para um enredo. Aqui está um enredo usando o ggplot2 biblioteca:
library(ggplot2)
qplot(ob_time, dist.mean, data=data, colour=groupname, geom=c("line","point")) +
geom_errorbar(aes(ymin=dist.mean-dist.sd, ymax=dist.mean+dist.sd))
Isso espanca os valores de tempo ao longo da escala natural, você pode usar scale_x_continuous Para definir os marcos de ticks nos valores de tempo real. Tê -los igualmente espaçados é mais complicado: você pode converter ob_time para um fator, mas então qplot se recusa a conectar os pontos com uma linha.
Solução 1 - Gráfico de barras:
qplot(factor(ob_time), dist.mean, data=data, geom=c("bar"), fill=groupname,
colour=groupname, position="dodge") +
geom_errorbar(aes(ymin=dist.mean-dist.sd, ymax=dist.mean+dist.sd), position="dodge")
Solução 2 - Adicione linhas manualmente usando o 1,2, ... Recodificação do fator:
qplot(factor(ob_time), dist.mean, data=data, geom=c("line","point"), colour=groupname) +
geom_errorbar(aes(ymin=dist.mean-dist.sd, ymax=dist.mean+dist.sd)) +
geom_line(aes(x=as.numeric(factor(ob_time))))
Outras dicas
Esta é uma dica da maneira como eu tentaria fazê -lo. Eu ignorei o agrupamento, então você terá que modificá -lo para incluir mais de uma série. Também não usei o zoológico porque não sei muito.
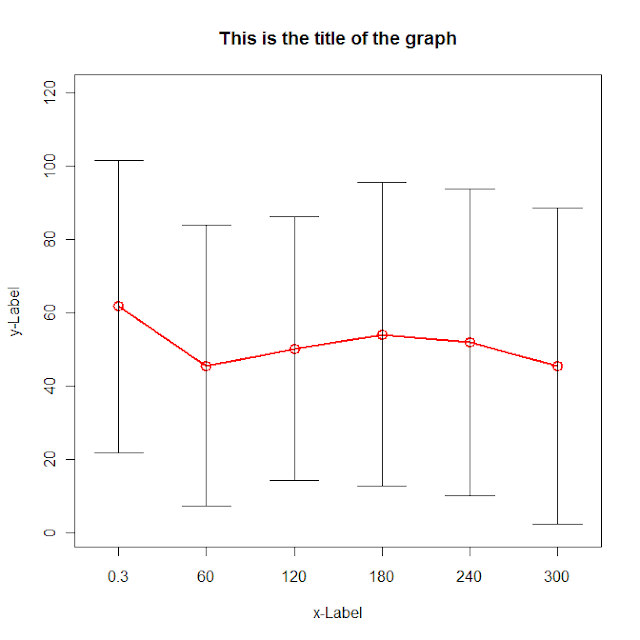
g <- (nrow(data)-1)/(3*nrow(data))
plot(data[,"dist.mean"],col=2, type='o',lwd=2,cex=1.5, main="This is the title of the graph",
xlab="x-Label", ylab="y-Label", xaxt="n",
ylim=c(0,max(data[,"dist.mean"])+max(data[,"dist.sd"])),
xlim=c(1-g,nrow(data)+g))
axis(side=1,at=c(1:nrow(data)),labels=data[,"ob_time"])
for (i in 1:nrow(data)) {
lines(c(i,i),c(data[i,"dist.mean"]+data[i,"dist.sd"],data[i,"dist.mean"]-data[i,"dist.sd"]))
lines(c(i-g,i+g),c(data[i,"dist.mean"]+data[i,"dist.sd"], data[i,"dist.mean"]+data[i,"dist.sd"]))
lines(c(i-g,i+g),c(data[i,"dist.mean"]-data[i,"dist.sd"], data[i,"dist.mean"]-data[i,"dist.sd"]))
}
Leia os dados no uso de read.zoo com o argumento split = para dividi -los por grupo. Em seguida, une as linhas distiadas, inferiores e superiores. Finalmente plote -os.
Lines <- "groupname ob_time dist.mean dist.sd dur.mean dur.sd ct.mean ct.sd
rowA 0.3 61.67500 39.76515 43.67500 26.35027 8.666667 11.29226
rowA 60.0 45.49167 38.30301 37.58333 27.98207 8.750000 12.46176
rowA 120.0 50.22500 35.89708 40.40000 24.93399 8.000000 10.23363
rowA 180.0 54.05000 41.43919 37.98333 28.03562 8.750000 11.97061
rowB 240.0 51.97500 41.75498 35.60000 25.68243 28.583333 46.14692
rowB 300.0 45.50833 43.10160 32.20833 27.37990 12.833333 14.21800"
library(zoo)
# next line is only needed until next version of zoo is released
source("http://r-forge.r-project.org/scm/viewvc.php/*checkout*/pkg/zoo/R/read.zoo.R?revision=719&root=zoo")
z <- read.zoo(textConnection(Lines), header = TRUE, split = 1, index = 2)
# pick out the dist and sd columns binding dist with lower & upper
z.dist <- z[, grep("dist.mean", colnames(z))]
z.sd <- z[, grep("dist.sd", colnames(z))]
zz <- cbind(z = z.dist, lower = z.dist - z.sd, upper = z.dist + z.sd)
# plot using N panels
N <- ncol(z.dist)
ylab <- sub("dist.mean.", "", colnames(z.dist))
plot(zz, screen = 1:N, type = "l", lty = rep(1:2, N*1:2), ylab = ylab)
Eu não acho que você precisa criar objetos de zoológico para esse tipo de gráfico, eu o faria diretamente no quadro de dados. Obviamente, pode haver outras razões para usar objetos de zoológico, uma fusão, agregação tão inteligente, etc.
Uma opção é o segplot Função de LatticeExtra
library(latticeExtra)
segplot(ob_time ~ (dist.mean + dist.sd) + (dist.mean - dist.sd) | groupname,
data = data, centers = dist.mean, horizontal = FALSE)
## and with the latest version of latticeExtra (from R-forge):
trellis.last.object(segments.fun = panel.arrows, ends = "both", angle = 90, length = .1) +
xyplot(dist.mean ~ ob_time | groupname, data, col = "black", type = "l")
Usando o conjunto de dados bem-reproduzível de Gabor, isso produz:
