utilizzando R.zoo per tracciare più serie con barre di errore
-
26-09-2019 - |
Domanda
Ho dati che assomiglia a questo:
> head(data)
groupname ob_time dist.mean dist.sd dur.mean dur.sd ct.mean ct.sd
1 rowA 0.3 61.67500 39.76515 43.67500 26.35027 8.666667 11.29226
2 rowA 60.0 45.49167 38.30301 37.58333 27.98207 8.750000 12.46176
3 rowA 120.0 50.22500 35.89708 40.40000 24.93399 8.000000 10.23363
4 rowA 180.0 54.05000 41.43919 37.98333 28.03562 8.750000 11.97061
5 rowA 240.0 51.97500 41.75498 35.60000 25.68243 28.583333 46.14692
6 rowA 300.0 45.50833 43.10160 32.20833 27.37990 12.833333 14.21800
Ogni nomegruppo è una serie di dati. Dal momento che voglio tracciare ogni serie a parte, li ho separato in questo modo:
> A <- zoo(data[which(groupname=='rowA'),3:8],data[which(groupname=='rowA'),2])
> B <- zoo(data[which(groupname=='rowB'),3:8],data[which(groupname=='rowB'),2])
> C <- zoo(data[which(groupname=='rowC'),3:8],data[which(groupname=='rowC'),2])
ETA:
Thanks to gd047: Now I'm using this:
z <- dlply(data,.(groupname),function(x) zoo(x[,3:8],x[,2]))
Gli oggetti zoo risultanti simile a questa:
> head(z$rowA)
dist.mean dist.sd dur.mean dur.sd ct.mean ct.sd
0.3 61.67500 39.76515 43.67500 26.35027 8.666667 11.29226
60 45.49167 38.30301 37.58333 27.98207 8.750000 12.46176
120 50.22500 35.89708 40.40000 24.93399 8.000000 10.23363
180 54.05000 41.43919 37.98333 28.03562 8.750000 11.97061
240 51.97500 41.75498 35.60000 25.68243 28.583333 46.14692
300 45.50833 43.10160 32.20833 27.37990 12.833333 14.21800
Quindi, se voglio tracciare dist.mean contro il tempo e includo barre di errore pari a +/- dist.sd per ogni serie:
- Come faccio a combinare A, B, C e dist.mean dist.sd?
- come faccio a fare un grafico a barre, o forse meglio , un grafico a linee dell'oggetto risultante?
Soluzione
Non vedere il punto di rottura i dati in tre pezzi solo per avere di combinare insieme per una trama. Ecco un grafico che utilizza la libreria ggplot2:
library(ggplot2)
qplot(ob_time, dist.mean, data=data, colour=groupname, geom=c("line","point")) +
geom_errorbar(aes(ymin=dist.mean-dist.sd, ymax=dist.mean+dist.sd))
Questa spazi i valori di tempo lungo la scala naturale, è possibile utilizzare scale_x_continuous per definire i segni di graduazione ai valori di tempo effettivo. Averli equidistanti è più complicato:. È possibile convertire ob_time ad un fattore, ma poi qplot rifiuta di collegare i punti con una linea
Soluzione 1 - grafico a barre:
qplot(factor(ob_time), dist.mean, data=data, geom=c("bar"), fill=groupname,
colour=groupname, position="dodge") +
geom_errorbar(aes(ymin=dist.mean-dist.sd, ymax=dist.mean+dist.sd), position="dodge")
Soluzione 2 - linee aggiuntivi utilizzando manualmente il 1,2, ... ricodifica del fattore:
qplot(factor(ob_time), dist.mean, data=data, geom=c("line","point"), colour=groupname) +
geom_errorbar(aes(ymin=dist.mean-dist.sd, ymax=dist.mean+dist.sd)) +
geom_line(aes(x=as.numeric(factor(ob_time))))
Altri suggerimenti
Questo è un suggerimento del modo in cui vorrei provare a farlo. Ho ignorato raggruppamento, quindi dovrete modificarlo per includere più di una serie. Anche io non ho usato zoo perché io non ne so molto.
g <- (nrow(data)-1)/(3*nrow(data))
plot(data[,"dist.mean"],col=2, type='o',lwd=2,cex=1.5, main="This is the title of the graph",
xlab="x-Label", ylab="y-Label", xaxt="n",
ylim=c(0,max(data[,"dist.mean"])+max(data[,"dist.sd"])),
xlim=c(1-g,nrow(data)+g))
axis(side=1,at=c(1:nrow(data)),labels=data[,"ob_time"])
for (i in 1:nrow(data)) {
lines(c(i,i),c(data[i,"dist.mean"]+data[i,"dist.sd"],data[i,"dist.mean"]-data[i,"dist.sd"]))
lines(c(i-g,i+g),c(data[i,"dist.mean"]+data[i,"dist.sd"], data[i,"dist.mean"]+data[i,"dist.sd"]))
lines(c(i-g,i+g),c(data[i,"dist.mean"]-data[i,"dist.sd"], data[i,"dist.mean"]-data[i,"dist.sd"]))
}
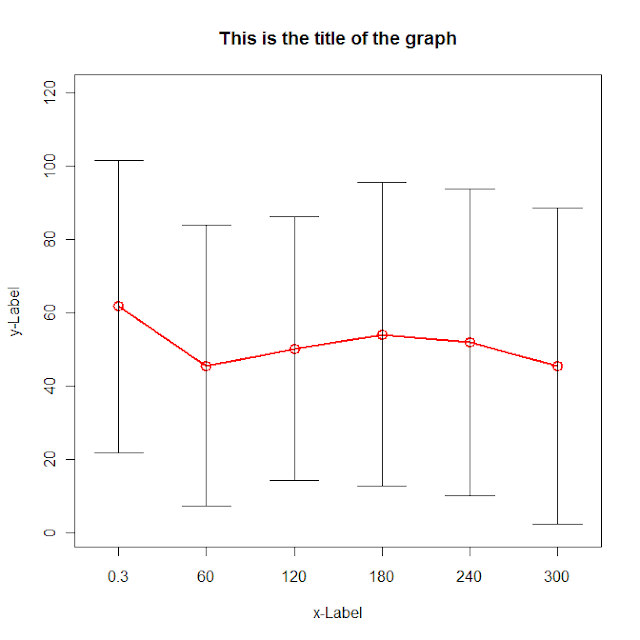
Leggi i dati in uso read.zoo con la scissione = argomento per dividerlo dalla nomegruppo. Poi si legano insieme la dist, inferiore e linee superiori. Infine tracciarli.
Lines <- "groupname ob_time dist.mean dist.sd dur.mean dur.sd ct.mean ct.sd
rowA 0.3 61.67500 39.76515 43.67500 26.35027 8.666667 11.29226
rowA 60.0 45.49167 38.30301 37.58333 27.98207 8.750000 12.46176
rowA 120.0 50.22500 35.89708 40.40000 24.93399 8.000000 10.23363
rowA 180.0 54.05000 41.43919 37.98333 28.03562 8.750000 11.97061
rowB 240.0 51.97500 41.75498 35.60000 25.68243 28.583333 46.14692
rowB 300.0 45.50833 43.10160 32.20833 27.37990 12.833333 14.21800"
library(zoo)
# next line is only needed until next version of zoo is released
source("http://r-forge.r-project.org/scm/viewvc.php/*checkout*/pkg/zoo/R/read.zoo.R?revision=719&root=zoo")
z <- read.zoo(textConnection(Lines), header = TRUE, split = 1, index = 2)
# pick out the dist and sd columns binding dist with lower & upper
z.dist <- z[, grep("dist.mean", colnames(z))]
z.sd <- z[, grep("dist.sd", colnames(z))]
zz <- cbind(z = z.dist, lower = z.dist - z.sd, upper = z.dist + z.sd)
# plot using N panels
N <- ncol(z.dist)
ylab <- sub("dist.mean.", "", colnames(z.dist))
plot(zz, screen = 1:N, type = "l", lty = rep(1:2, N*1:2), ylab = ylab)
Non credo che è necessario creare oggetti zoo per questo tipo di trama, lo farei direttamente dal frame di dati. Naturalmente, ci possono essere altri motivi per oggetti uso zoo, una fusione come intelligente, l'aggregazione, ecc.
Una possibilità è la funzione segplot da latticeExtra
library(latticeExtra)
segplot(ob_time ~ (dist.mean + dist.sd) + (dist.mean - dist.sd) | groupname,
data = data, centers = dist.mean, horizontal = FALSE)
## and with the latest version of latticeExtra (from R-forge):
trellis.last.object(segments.fun = panel.arrows, ends = "both", angle = 90, length = .1) +
xyplot(dist.mean ~ ob_time | groupname, data, col = "black", type = "l")
Utilizzo di dati ben riproducibile di Gabor questo produce:
