mit R.zoo auf mehrere Serien mit Fehlerbalken plotten
-
26-09-2019 - |
Frage
Ich habe Daten, die wie folgt aussieht:
> head(data)
groupname ob_time dist.mean dist.sd dur.mean dur.sd ct.mean ct.sd
1 rowA 0.3 61.67500 39.76515 43.67500 26.35027 8.666667 11.29226
2 rowA 60.0 45.49167 38.30301 37.58333 27.98207 8.750000 12.46176
3 rowA 120.0 50.22500 35.89708 40.40000 24.93399 8.000000 10.23363
4 rowA 180.0 54.05000 41.43919 37.98333 28.03562 8.750000 11.97061
5 rowA 240.0 51.97500 41.75498 35.60000 25.68243 28.583333 46.14692
6 rowA 300.0 45.50833 43.10160 32.20833 27.37990 12.833333 14.21800
Jeder Gruppenname ist eine Datenreihe. Da ich jede Reihe separat dargestellt werden soll, habe ich sie wie folgt getrennt:
> A <- zoo(data[which(groupname=='rowA'),3:8],data[which(groupname=='rowA'),2])
> B <- zoo(data[which(groupname=='rowB'),3:8],data[which(groupname=='rowB'),2])
> C <- zoo(data[which(groupname=='rowC'),3:8],data[which(groupname=='rowC'),2])
ETA:
Thanks to gd047: Now I'm using this:
z <- dlply(data,.(groupname),function(x) zoo(x[,3:8],x[,2]))
Die resultierenden Zoo-Objekte wie folgt aussehen:
> head(z$rowA)
dist.mean dist.sd dur.mean dur.sd ct.mean ct.sd
0.3 61.67500 39.76515 43.67500 26.35027 8.666667 11.29226
60 45.49167 38.30301 37.58333 27.98207 8.750000 12.46176
120 50.22500 35.89708 40.40000 24.93399 8.000000 10.23363
180 54.05000 41.43919 37.98333 28.03562 8.750000 11.97061
240 51.97500 41.75498 35.60000 25.68243 28.583333 46.14692
300 45.50833 43.10160 32.20833 27.37990 12.833333 14.21800
Wenn ich also dist.mean gegen die Zeit darstellen möchten und beinhalten Fehlerbalken gleich zu +/- dist.sd für jede Serie:
- Wie kombiniere ich A, B, C dist.mean und dist.sd?
- Wie mache ich ein Balkendiagramm, oder vielleicht besser , ein Liniendiagramm des resultierenden Objekts?
Lösung
Ich sehe nicht den Punkt bricht die Daten in drei Stücke nur bis hat es für eine Handlung zusammen zu kombinieren. Hier ist eine Darstellung, die die ggplot2 Bibliothek:
library(ggplot2)
qplot(ob_time, dist.mean, data=data, colour=groupname, geom=c("line","point")) +
geom_errorbar(aes(ymin=dist.mean-dist.sd, ymax=dist.mean+dist.sd))
Diese Bereiche sind die Zeitwerte entlang der natürlichen Skala, können Sie scale_x_continuous verwenden, um die Skalenstriche an den tatsächlichen Zeitwerte zu definieren. sie mit gleichem Abstand zu haben, ist kniffliger. Sie können ob_time zu einem Faktor konvertieren, aber dann qplot weigert sich, die Punkte mit einer Linie zu verbinden
Lösung 1 - Bargraph:
qplot(factor(ob_time), dist.mean, data=data, geom=c("bar"), fill=groupname,
colour=groupname, position="dodge") +
geom_errorbar(aes(ymin=dist.mean-dist.sd, ymax=dist.mean+dist.sd), position="dodge")
Lösung 2 - Add Linien manuell die 1,2 verwenden, ... Umkodierung des Faktors:
qplot(factor(ob_time), dist.mean, data=data, geom=c("line","point"), colour=groupname) +
geom_errorbar(aes(ymin=dist.mean-dist.sd, ymax=dist.mean+dist.sd)) +
geom_line(aes(x=as.numeric(factor(ob_time))))
Andere Tipps
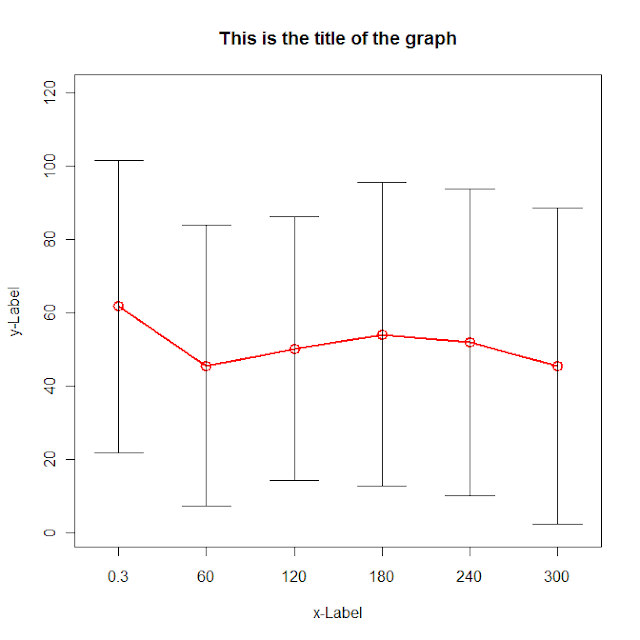
Dies ist ein Hinweis auf die Art, wie ich versuchen würde, es zu tun. Ich habe Gruppierung ignoriert, so dass Sie es ändern müssen, werden mehr als eine Serie aufzunehmen. Ich habe auch nicht verwendet Zoo, weil ich nicht viel wissen.
g <- (nrow(data)-1)/(3*nrow(data))
plot(data[,"dist.mean"],col=2, type='o',lwd=2,cex=1.5, main="This is the title of the graph",
xlab="x-Label", ylab="y-Label", xaxt="n",
ylim=c(0,max(data[,"dist.mean"])+max(data[,"dist.sd"])),
xlim=c(1-g,nrow(data)+g))
axis(side=1,at=c(1:nrow(data)),labels=data[,"ob_time"])
for (i in 1:nrow(data)) {
lines(c(i,i),c(data[i,"dist.mean"]+data[i,"dist.sd"],data[i,"dist.mean"]-data[i,"dist.sd"]))
lines(c(i-g,i+g),c(data[i,"dist.mean"]+data[i,"dist.sd"], data[i,"dist.mean"]+data[i,"dist.sd"]))
lines(c(i-g,i+g),c(data[i,"dist.mean"]-data[i,"dist.sd"], data[i,"dist.mean"]-data[i,"dist.sd"]))
}
Lesen Sie die Daten bei der Nutzung read.zoo mit dem geteilten = Argumente durch Gruppennamen zu spalten. Dann binden die zusammen dist, untere und obere Linien. Schließlich sie zeichnen.
Lines <- "groupname ob_time dist.mean dist.sd dur.mean dur.sd ct.mean ct.sd
rowA 0.3 61.67500 39.76515 43.67500 26.35027 8.666667 11.29226
rowA 60.0 45.49167 38.30301 37.58333 27.98207 8.750000 12.46176
rowA 120.0 50.22500 35.89708 40.40000 24.93399 8.000000 10.23363
rowA 180.0 54.05000 41.43919 37.98333 28.03562 8.750000 11.97061
rowB 240.0 51.97500 41.75498 35.60000 25.68243 28.583333 46.14692
rowB 300.0 45.50833 43.10160 32.20833 27.37990 12.833333 14.21800"
library(zoo)
# next line is only needed until next version of zoo is released
source("http://r-forge.r-project.org/scm/viewvc.php/*checkout*/pkg/zoo/R/read.zoo.R?revision=719&root=zoo")
z <- read.zoo(textConnection(Lines), header = TRUE, split = 1, index = 2)
# pick out the dist and sd columns binding dist with lower & upper
z.dist <- z[, grep("dist.mean", colnames(z))]
z.sd <- z[, grep("dist.sd", colnames(z))]
zz <- cbind(z = z.dist, lower = z.dist - z.sd, upper = z.dist + z.sd)
# plot using N panels
N <- ncol(z.dist)
ylab <- sub("dist.mean.", "", colnames(z.dist))
plot(zz, screen = 1:N, type = "l", lty = rep(1:2, N*1:2), ylab = ylab)
Ich glaube nicht, Sie Zoo-Objekte für diese Art von Handlung schaffen müssen, würde ich es aus dem Datenrahmen direkt tun. Natürlich kann es andere Gründe geben Verwendung Zoo Objekte, wie eine intelligente Verschmelzung, Aggregation, etc.
Eine Option ist die segplot Funktion von latticeExtra
library(latticeExtra)
segplot(ob_time ~ (dist.mean + dist.sd) + (dist.mean - dist.sd) | groupname,
data = data, centers = dist.mean, horizontal = FALSE)
## and with the latest version of latticeExtra (from R-forge):
trellis.last.object(segments.fun = panel.arrows, ends = "both", angle = 90, length = .1) +
xyplot(dist.mean ~ ob_time | groupname, data, col = "black", type = "l")
Mit der Gabor gut reproduzierbaren-Datensatz erzeugt dies:
